Appendix 1: Visualization¶
本節では、Jupyter Notebook上での原子系の可視化方法に関するテクニックを紹介します。
目次¶
ASE
atomsのpng画像保存
trajのgif画像保存
nglviewer
ASE + nglviewer での表示。
pfcc-extrasの使い方
view_ngl関数のそれぞれの引数の説明
Tooltipで原子番号やposition表示
png, html での保存に対応
show charge (要Calculator)
show force (要Calculator)
nglviewer tips
右クリックで距離・角度・2面角表示
povray
povrayを用いたgif画像描画
他の可視化ツール
VMD, OVITOなどでの表示
事前準備¶
ここでは、1つの系 atoms を可視化する場合と、時系列である traj を表示する場合を紹介します。
まずは、今回可視化するatoms と traj を用意します。
[1]:
from ase.build import bulk
atoms = bulk("Si", cubic=True) * (3, 3, 3)
from ase.visualize import view
view(atoms, viewer="ngl")
[1]:
[2]:
from pfp_api_client.pfp.calculators.ase_calculator import ASECalculator
from pfp_api_client.pfp.estimator import Estimator, EstimatorCalcMode
estimator = Estimator(calc_mode=EstimatorCalcMode.PBE, model_version="v8.0.0")
calculator = ASECalculator(estimator)
[3]:
from ase.md.velocitydistribution import MaxwellBoltzmannDistribution, Stationary
from ase.md.verlet import VelocityVerlet
from ase.io import Trajectory
from ase import units
atoms.calc = calculator
# Set the momenta corresponding to T=500K.
MaxwellBoltzmannDistribution(atoms, temperature_K=5000.0)
# Sets the center-of-mass momentum to zero.
Stationary(atoms)
# Run MD using the VelocityVerlet algorithm
dyn = VelocityVerlet(atoms, 1.0 * units.fs, trajectory="output/dyn.traj")
def print_dyn():
print(f"Dyn step: {dyn.get_number_of_steps(): >3}, energy: {atoms.get_total_energy():.3f}")
dyn.attach(print_dyn, interval=10)
dyn.run(100)
Dyn step: 0, energy: -847.935
Dyn step: 10, energy: -847.842
Dyn step: 20, energy: -847.837
Dyn step: 30, energy: -847.867
Dyn step: 40, energy: -847.854
Dyn step: 50, energy: -847.846
Dyn step: 60, energy: -847.849
Dyn step: 70, energy: -847.847
Dyn step: 80, energy: -847.850
Dyn step: 90, energy: -847.852
Dyn step: 100, energy: -847.844
[3]:
True
ASE¶
まずはじめに、ASE組み込みの機能を用いたpng画像やgif アニメーションの作成方法を紹介します。
write methodを用いて atoms の場合は “.png” 、 traj の場合は “.gif” の拡張子を指定することでそれぞれの形式での可視化・ファイル保存が可能です。 保存されたファイルは、たとえば IPython.display.Image などを用いるとJupyter Notebook上で可視化可能です。
[4]:
from ase.io import write
from IPython.display import Image
write("output/Si.png", atoms, rotation="0x,0y,0z")
Image(url="output/Si.png", width=300)
[4]:
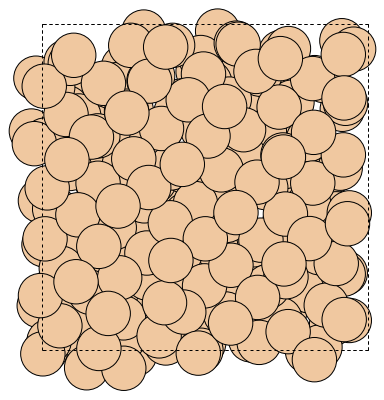
rotation の値を変えることで表示する系を回転させることができます。
[5]:
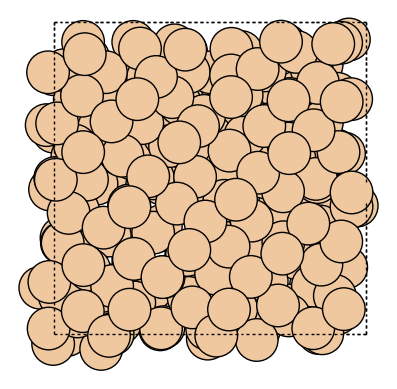
write("output/Si_rotate_view.png", atoms, rotation="30x,30y,30z")
Image(url="output/Si_rotate_view.png", width=300)
[5]:

複数の atoms = Trajectory traj をアニメーションにしたい場合は、 “.gif” 拡張子を指定することで gif動画にできます。
[6]:
traj = Trajectory("output/dyn.traj")
write("output/Si.gif", traj[::10], rotation="0x,0y,0z")
[7]:
Image(url="output/Si.gif", width=300)
[7]:

matplotlibを経由した描画や、ファイル保存も以下のようにできます。
[8]:
import matplotlib.pyplot as plt
from ase.visualize.plot import plot_atoms
fig, ax = plt.subplots()
plot_atoms(atoms, ax, radii=0.3, rotation=("0x,0y,0z"))
fig.savefig("ase_slab.png")
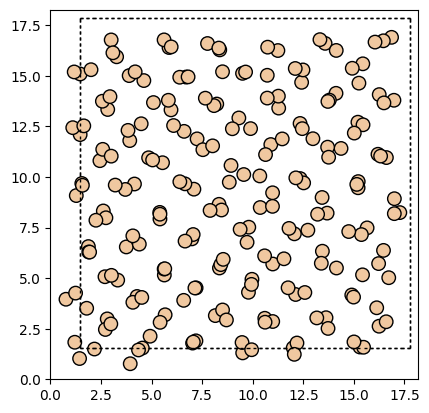
その他、ASEの可視化関数に関する詳細は以下を参照ください。
nglviewer¶
nglview というライブラリをインストールすることで、3次元の系をInteractiveに動かせるように表示することも可能です。
いくつか方法があります。
方法1. nglview提供の機能を使う方法¶
atoms の表示は show_ase、traj の表示は show_asetraj methodを用いることで表示が可能です。
gui=True のOptionを加えると、下部にいろいろな設定を変えられるタブが表示されます。
[9]:
import nglview as nv
nv.show_ase(atoms, gui=True)
[10]:
nv.show_asetraj(traj[::10], gui=True)
gui_style で表示形式を変更することもできます。
[11]:
# v = nv.show_asetraj(traj[::10], gui=False)
# v.gui_style = "ngl"
# v
上記を実行すると、以下のような画面が出ます。
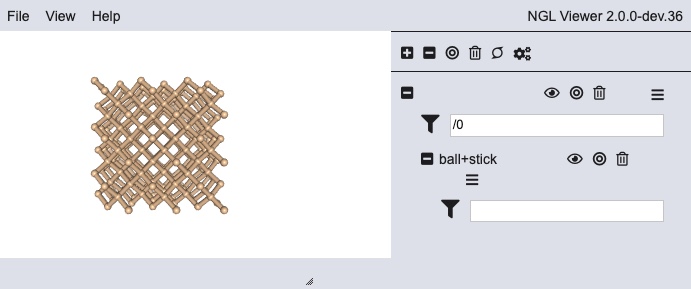
ただし、この表示を行うとHTML でのdocument表示時にcssスタイルが崩れる問題が知られているため、 ここではスクリーンショット表示としています。
[12]:
v = nv.show_asetraj(traj[::10], gui=False)
v.add_representation("ball+stick")
v
方法2. ASEのViewerを経由して、nglviewを用いる方法¶
ASE の view method に viewer="ngl" を指定することでも、表示が可能です。 ASE のViewerでは右側にいくつかの設定値を変えられるOptionが出てきます。
[13]:
from ase.visualize import view
view(atoms, viewer="ngl")
[13]:
Trajectory 表示の場合も同じ view methodが使えます。
[14]:
view(traj[::10], viewer="ngl")
[14]:
pfcc-extras¶
Matlantis内で提供されているpfcc-extrasライブラリにて、さらにカスタマイズを加えたviewer を提供しています。
view_ngl method を用いて可視化が可能で、以下のような設定ができます。
representations: NGL Viewer固有のRepresentationsが指定できます。特に使う機能としては、"ball+stick"を指定することで、分子の結合を表示する事ができます。w, h: NGL Viewerのwidth, height の指定ができます。
追加機能(一部):
Tooltipで原子番号やposition表示
png, html での保存に対応
show charge (要Calculator)
show force (要Calculator)
[15]:
from pfcc_extras.visualize.view import view_ngl
view_ngl(atoms, representations=["ball+stick"], w=400, h=300)
[15]:
Tooltipで原子番号やposition表示¶
各原子にマウスオーバーをした際に、原子番号や各原子の座標値が表示されるようにしています。
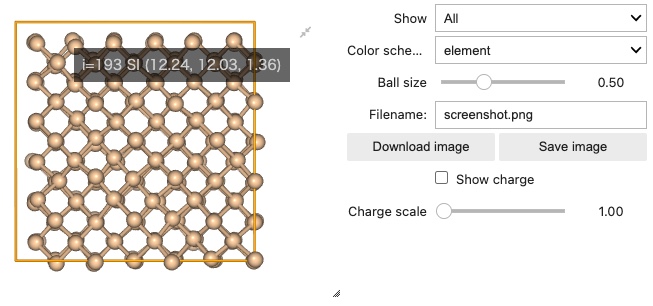
png, html 形式での保存に対応¶
“Download image” ボタンを押すと、今表示されている画像を手元のPCにダウンロードします。 “Save image” ボタンを押すと、今表示されている画像をカレントディレクトリに保存します。
デフォルトでは、“screenshot.png”となっていますが、拡張子を .html にすることで、HTML形式での保存も可能です。
Show force (要Calculator)¶
“Show force” にチェックを入れることで、各原子に働いている力 (force vector)の可視化ができます。 “Force scale” を変えることでベクトルの大きさを変えることができます。
※ atomsに対して calculator をセットしておく必要があります。
[16]:
from ase.build import molecule
ch3cho_atoms = molecule("CH3CHO")
ch3cho_atoms.pop(0)
ch3cho_atoms.calc = calculator
view_ngl(ch3cho_atoms, representations=["ball+stick"], w=400, h=300, show_force=True)
[16]:
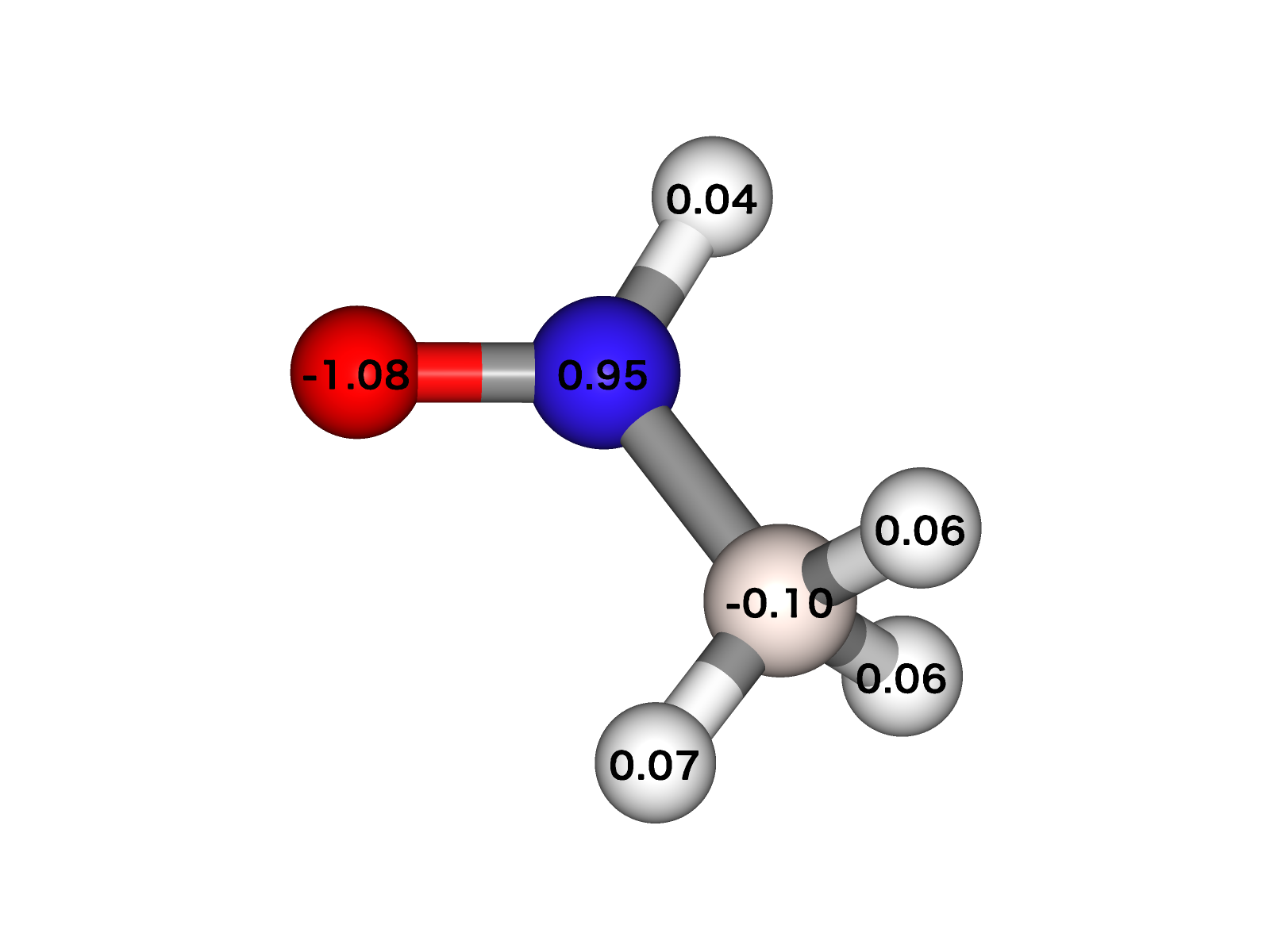
Show charge (要Calculator)¶
“Show charge” にチェックを入れることで、各原子のchargeの可視化ができます。 “Charge scale” を変えることで色の濃淡を変えることができます。
※ atomsに対して charge計算が可能なcalculator をセットしておく必要があります。
[17]:
from ase.build import molecule
ch3cho_atoms = molecule("CH3CHO")
ch3cho_atoms.calc = calculator
view_ngl(ch3cho_atoms, representations=["ball+stick"], w=400, h=300)
[17]:
view.add_label を用いることで、chargeの値を原子上に表示することも可能です。
[18]:
v = view_ngl(ch3cho_atoms, representations=["ball+stick"], w=400, h=300)
v.gui.show_charge_checkbox.value = True
v.view.add_label(
color="black", labelType="text",
labelText=[f"{charge:.2f}" for charge in ch3cho_atoms.get_charges().ravel()],
zOffset=1.0, attachment="middle_center", radius=0.5)
v
[18]:
表示例:
replace_structure¶
デフォルトのnglviewer では、Trajectoryを表示する際、一番はじめのAtoms に対してBond, Cell のみを設定して、Frameの移動時には座標値のみが更新されます。そのため、
Bondの変化 (切れたりつながったり)
Cell sizeの変化
原子数の増減 (Ex. LAMMPSでのEvapolateやDepositに相当する操作)
元素種の変化
などがあるようなTrajectoryは正しく表示できません。
以下の例のように、replace_structure=True を設定することでこれらのケースも表示することができます。 (ただし、毎回Bond 表示更新などの計算が走るため、少し動作は重くなります。)
[19]:
ch3cl_atoms = molecule("CH3Cl")
atoms_list = [ch3cl_atoms, ch3cho_atoms]
view_ngl(atoms_list, representations=["ball+stick"], replace_structure=True, w=400, h=300)
[19]:
nglviewer tips¶
右クリックでの距離・角度・2面角表示¶
NGLViewerでは、右クリックを押して原子を選択して、以下のような値を計測・表示させることが可能です。
距離: 原子1を1回右クリック -> 原子2を2回右クリック
角度: 原子1を1回右クリック -> 原子2を1回右クリック -> 原子3を2回右クリック
二面角: 原子1を1回右クリック -> 原子2を1回右クリック -> 原子3を1回右クリック -> 原子4を2回右クリック
[20]:
view_ngl(ch3cho_atoms, representations=["ball+stick"], replace_structure=True, w=400, h=300)
[20]:
例:
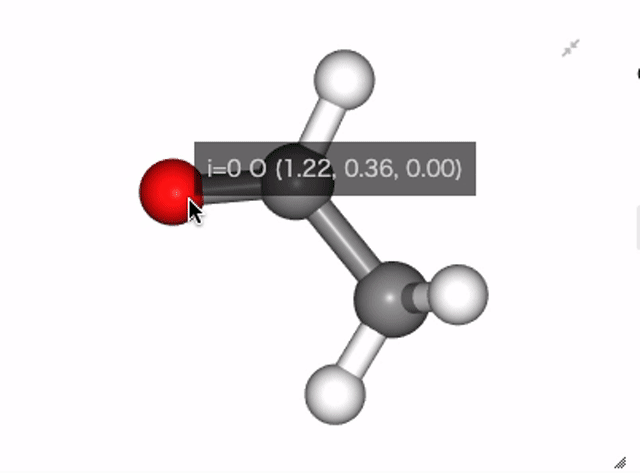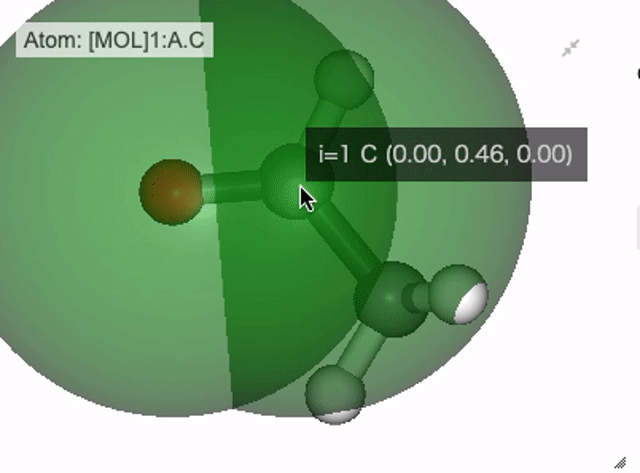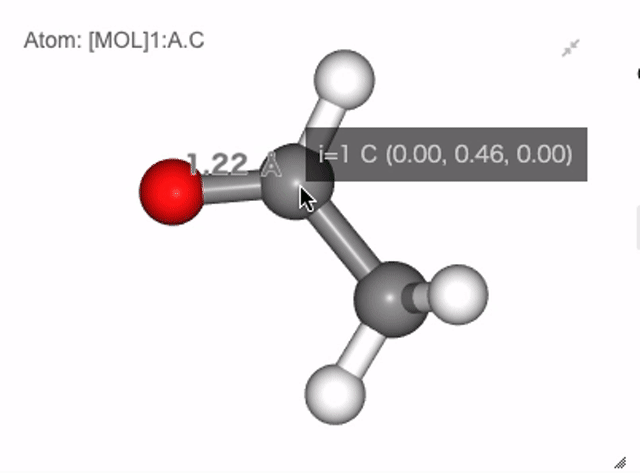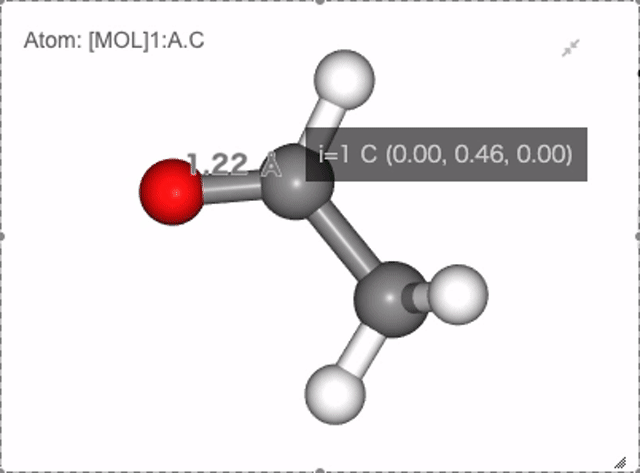
Show distance between atom 0 and 1
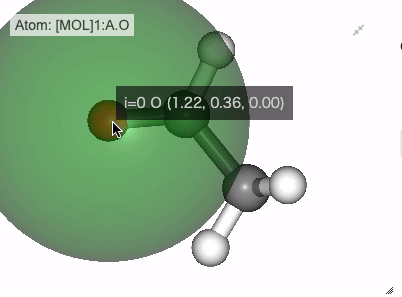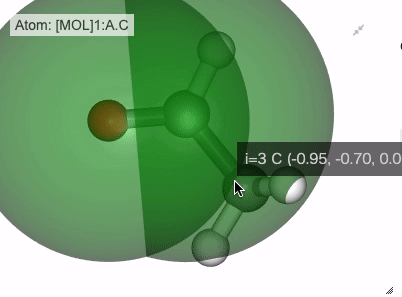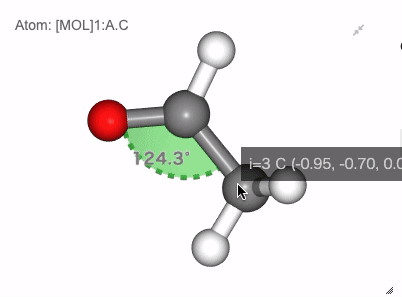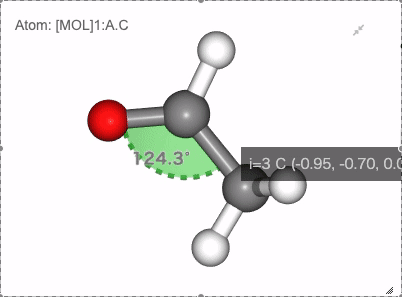
Show angle of atom 0, 1 and 2
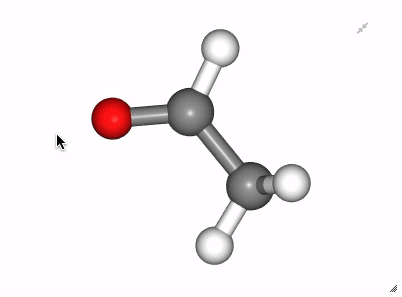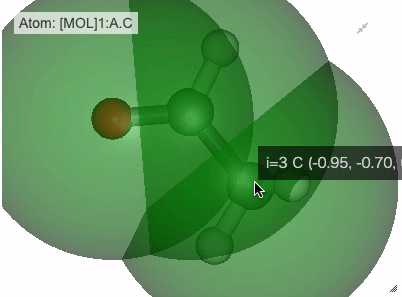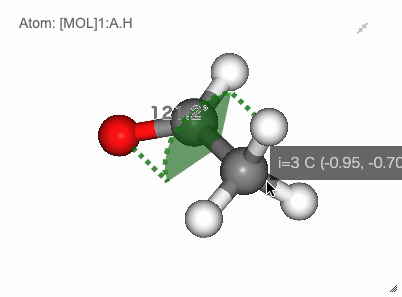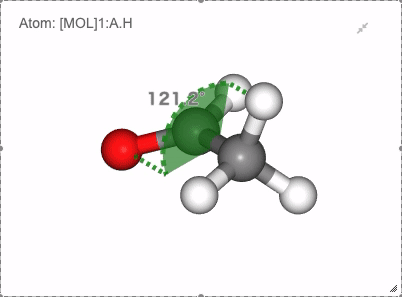
Show dihedral angle of atom 0, 1, 2 and 3
povray¶
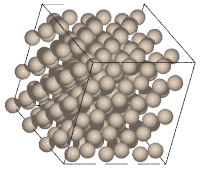
povray を利用して原子系を可視化することも可能です。
povrayをインストールし、povrayコマンドが使える環境で、以下の関数を実行することで、Trajectoryのgif画像作成ができます。
[21]:
from pfcc_extras.visualize.povray import traj_to_gif
traj_to_gif(
traj[::10],
gif_filepath="output/Si_anim.gif",
povdir="output/pov",
pngdir="output/png",
clean=False
)
[Parallel(n_jobs=4)]: Using backend ThreadingBackend with 4 concurrent workers.
[Parallel(n_jobs=4)]: Done 11 out of 11 | elapsed: 6.8s finished
[22]:
Image("output/Si_anim.gif")
[22]:
<IPython.core.display.Image object>
[23]:
from pfcc_extras.visualize.povray import traj_to_apng
traj_to_apng(
traj[::10],
apng_filepath="output/Si_anim.png",
povdir="output/pov",
pngdir="output/png",
clean=False
)
[Parallel(n_jobs=4)]: Using backend ThreadingBackend with 4 concurrent workers.
[Parallel(n_jobs=4)]: Done 11 out of 11 | elapsed: 6.8s finished
[24]:
Image("output/Si_anim.png")
[24]:
別の可視化ツールを使う¶
他の可視化ツールを使いたい場合、traj ファイルを別形式で保存します。 ここでは、xyz, pdbファイルへの変換を行なっています。これらのファイル形式は以下のツールで利用可能です。
単一の系の表示には、
また、Trajectoryの可視化には、
などを使用することができます。
[25]:
from ase.io import write
# traj = Trajectory("output/dyn.traj")
write("output/si_traj.xyz", traj)
write("output/si_traj.pdb", traj)